原理
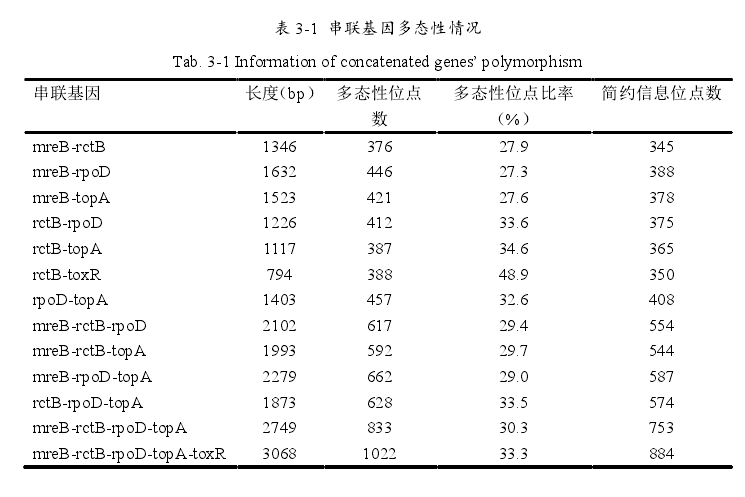
多位點序列分析(MLSA)方法不使用分配的等位基因,而是將持家基因的基因片段序列連線在一起,並使用此連線序列,以確定物種系統發育關係。多位點序列分析方法採用多個保守基因位點進行鑑定分型,保守基因的選擇對鑑定分型的結果至關重要。不同的基因由於進化速速率及進化時間的關係,有效的信息位點不一,其分辨力存在差異,為此,研究者創新性的將基因序列進行串聯,增加有效信息位點數量,以便提高分辨力對難以區分的相似菌群進行鑑定,這一方法被稱之為多位點序列分析(MLSA)。這一方法得益於
多位點序列分型方法的快速發展,是近些年用於鑑定爭議菌株的有力工具。多位點序列分析方法具有快速簡便、重複性好、高區分力的特點。同時,開發共享的多位點序列分析網際網路資料庫和高速發展的 PCR 測序技術優勢使該方法逐步被廣泛的套用於病原菌的鑑定分型研究中。
與
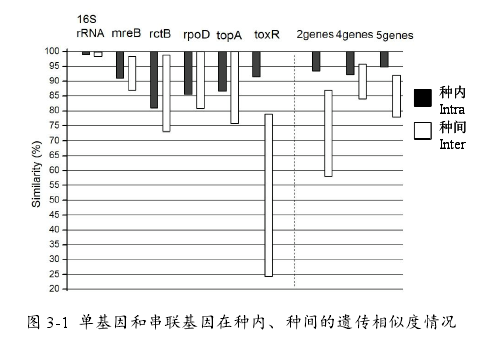
多位點序列分型不同,多位點序列分析法的序列之間的高相似性由單個核苷酸差異決定,序列之間的低相似性顯示多個核苷酸差異,因此,這種分析方法更加適用於具有克隆進化的生物體,不太適用於經常發生重組事件的生物。也可用來確定密切相關的物種之間的系統發育關係。
細菌基因在進化過程中的自然變異使得菌種鑑定研究者可以根據變異信息對菌株進行區分,然而不同的基因位點進化的速率有所差異,變異率大的基因能夠更好的區分菌種,然而細菌的快速繁殖及自由頻繁的信息交換使得基因獲得了更大的變異,一些基因位點的同源片段重組使得其辨別力降低,不能很好的鑑別發生同源重組的菌種,為此,研究者們在套用多位點序列分析法進行分析時,將用於鑑定分型的基因序列進行串聯拼接,增加了有效變異位點的數量,同時降低重組事件帶來的影響。
套用
多位點序列分析大量套用於研究不同的抗生素抵抗株、毒力或抗原的相關特殊基因型,以及新變異株引起的疾病流行病學研究。
2009年,Yoshizawa 等通過 MLSA 方法的分析發現,4 株分離自海水環境的發光弧菌在進化上歸為哈維弧菌類似群菌株,但是又形成兩個獨立的分支,研究者將其分別命名為 Vibrio azureus sp. nov.和 Vibrio sagamiensis sp. nov.。2010年,Cano 等利用 5 個基因(rpoA、pyrH、 topA、ftsZ、mreB)對 2 株分離自澳大利亞昆士蘭州養殖患病的白須龍蝦(Panulirus ornatus)和斑節對蝦(Penaeus monodon)的分離株進行分析發現,這兩株分離株區別於其它已知的哈維弧菌類似群菌株,並提議將其命為新種 V. owensii sp.nov.。2013年,Wang 等套用該方法成功的鑑定出 2011 年 5-7 月在福建漳浦養殖的凡納濱對蝦(Litopenaeus vannamei)暴發的弧菌發光病病原為坎氏弧菌。此外,研究者們通過多位點序列分析的方法分析已經發現分離自不同海洋深度的海綿弧菌分離株與近海岸的弧菌分離株是一個存在基因重組的隨機交流群體。
2014年,
上海海洋大學馮娟實驗室利用多位點序列分析法對採集自 2007-2012 年我國南方沿海地區患病海水魚的哈維弧菌分離株進行鑑定分型,為該類似群菌株引起的病害防治及流行病學調查提供了研究基礎,研究了多基因位點序列對哈維弧菌類似群的區分力及哈維弧菌分離株多基因位點分型情況,證明多位點序列分析是區分具有表型和基因型高相似度的哈維類似群菌株的有效方法。此研究也發現,最新命名的哈維弧菌類似群成員 V. communis為首次在國內被發現為海水魚病原菌,該兩株菌分別採集自 2011 年廣東流沙灣養殖的籃子魚(149DEJ)和卵形鯧鰺(155DEI)。