基本介紹
- 外文名:DGGE
- 性質:一種很有用分子標記方法
基本原理,特點,主要步驟,
基本原理
DGGE不是將分子量不同的DNA分開,而是通過聚丙烯醯胺凝膠中變性劑濃度梯度的不同,將序列不同的DNA分開。該方法的原理是根據DNA 的解鏈特性,不同鹼基組成的DNA雙螺旋發生變性所要求的變性劑濃度不同,混合雙鏈DNA在變性劑濃度呈線性梯度增加的聚丙烯醯胺凝膠電泳時,當泳動到與DNA變性所需變性劑濃度一致的凝膠位置時,相對應的DNA發生解鏈變性,導致電泳遷移速率降低。由於泳動受阻DNA分子在凝膠中的停留位置不同,從而使不同DNA分子得以分離。根據變性劑梯度方向的不同, DGGE可分為:垂直DGGE,即變性劑梯度與電場方向垂直,常用於試驗決定分離型、野生型和變異型的最佳變性劑梯度範圍;平行DGGE,其變性劑的梯度和電場方向平行,主要用於解鏈範圍明確的DNA片段的檢測。
特點
DGGE/TGGE已廣泛用於分析自然環境中細菌、藍細菌, 古菌、微型真核生物、真核生物和病毒群落的生物多樣性。這一技術能夠提供群落中優勢種類信息並同時分析多個樣品,具有可重複和操作簡單等特點, 適合於調查種群的時空變化, 並且可通過對條帶的序列分析或與特異性探針雜交分析鑑定群落組成。DGGE和TGGE分別通過逐漸增加的化學變性劑線性濃度梯度和線性溫度梯度可以把長度相同但只有一個鹼基不同的DNA片段分離。DNA分子的雙鏈在特定溫度下會分離,這個溫度取決於互補鏈的氫鍵含量(富含GC的區域融解溫度較高)和相鄰鹼基的引力。 DGGE系統
DGGE系統
 DGGE系統
DGGE系統主要步驟
DGGE對微生態的分析一般包括三個步驟:核酸提取,16SrRNA序列的PCR擴增以及DGGE指紋圖譜分析。有些學者通過克隆、測序建立微生物區系的16SrRNA/DNA文庫,通過系統發育分析,建立進化樹,從而獲得微生物多樣性信息,但這種方法相對於指紋圖譜技術來說,費時費力,價格也相對昂貴。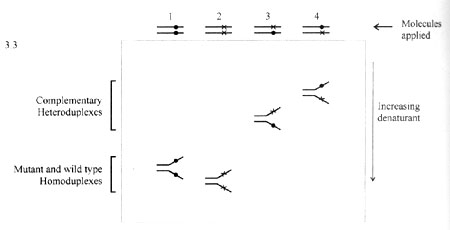

核酸提取
微生物總DNA的提取是整個分子生物學技術的基礎,是否能獲得具有代表性的總DNA樣品將決定後續分析的可行性。一般認為微生物提取總DNA的方法分為細胞裂解和核酸抽提兩個步驟。細胞裂解有三種:機械法、化學法和酶法。機械法包括玻璃珠法、超音波法、凍融法等;化學法包括加入SDS、苯酚等;酶法包括加入裂解酶、蛋白酶K 和溶解酶等。而許多研究者用其中兩種或三種方法進行組合,發現組合後提取總DNA 的效果較好。Stahl等(1988)在含有SDS和酚的體系中加入適量的玻璃微珠,機械法裂解瘤胃樣品中的菌體細胞。Zhu等用酶法和玻璃珠相結合的方法提取雞盲腸內容物的微生物總DNA。核酸抽提一般用酚- 氯仿-異戊醇抽提,這種方法對去除雜蛋白有良好的效果。Reichardt等(1994)認為, CTAB(十六烷基三甲基溴化胺)法也是常用的方法。不需要貴重儀器,操作較為簡單,能廣泛適用於各種植物的核酸提取,純度也能滿足許多分子生物學操作的要求,所以使用日趨廣泛。
 DNA螺旋
DNA螺旋

 DNA螺旋
DNA螺旋16SrRNA基因序列的PCR擴增
細菌的核糖體RNA按沉降係數分為5S、16S和23S三種,16SrRNA基因是細菌染色體上編碼該rRNA的相應DNA系列。16SrRNA基因序列全長1540bp ,有保守區和可變區之分,每種細菌的保守區都是相同的,能反映生物種類的親緣關係,為系統發育重建提供線索;可變區因細菌種類而異,通過保守區設計引物,擴增出可變區,就可以得知微生物多樣性信息。大多數情況下,可根據16SrRNA基因V3區和V6-V8區設計細菌特異性引物進行PCR擴增,有時為了更加準確得獲得微生物多樣性的信息,可先通過細菌16SrRNA序列全長通用引物進行PCR擴增,再對V3或V6-V8區進行擴增。Yu等的研究表明,通常使用16S的V3區進行PCR擴增後進行DGGE分析,如果所測的序列長度比較長時,也可以使用16S的V3-V5或V6-V8區。

DGGE指紋圖譜分析

