簡介
生物大分子三維空間結構資料庫是一類重要的生物信息學資料庫。蛋白質結構資料庫(ProreinData Bank,PDB)是1971年創建的國際上最著名、最完整的
蛋白質三維結構資料庫。另外還有蛋白質分類資料庫SCOP和CATH。SCOP是
英國醫學研究委員會分子生物學實驗室和蛋白質工程中心開發的基於Web的蛋白質結構分類、檢索和分析系統。CATH是另一個著名的蛋白質分類資料庫,由
英國倫敦大學開發和維護。
理論依據
20世紀60年代美國的Anfinsend小組作了
牛胰核糖核酸酶變性及復現再摺疊的實驗和理論研究,提出蛋白質的胺基酸序列包含有足夠的信息去決定它的空間結構(並因此獲得
諾貝爾獎),為由胺基酸序列預測蛋白質的三維空間結構建立了實驗和理論基礎。
蛋白質結構
蛋白質結構是發揮其生物學功能的基礎,儘管實驗方法可以解析出一部分蛋白質的高解析度結構,但結構預測的理論方法依然非常重要,它將為大部分蛋白的結構研究提供非常有價值的信息,尤其是對那些不能從實驗上測定結構的蛋白。實際上,精確的蛋白質
三維結構理論預測是困難的,現階段比較有效的途徑是先研究粗粒化結構,如蛋白質結構型和拓撲結構等。由英國倫敦大學UCL開發和維護的
蛋白質結構分類資料庫CATH(Class—Architecture.Topology-Homology)、由英國醫學研究委員會(
MRC)的分子生物學實驗室和蛋白質工程研究中心開發和維護蛋白質結構分類資料庫SCOP(Structural Classification OfProteins)都是圍繞粗粒化結構建立的。
蛋白質結構預測
套用PDB
對PDB資料庫庫進行數據挖掘和統計的最終目的是為了能夠通過對已知結構的蛋白質數據的數據挖掘,發現蛋白質序列和結構之間的某種聯繫或者規律,加深我們對蛋白質序列決定結構的機理的了解,並最終能尋找更好的蛋白質二級結構預測方法。
發展歷史
世界上第一個蛋白質晶體結構的測定和解析發生在50年代末60年代初。蛋白質二級結構預測工作開始於60年代中期,也就是說在解析出第一個蛋白質的三維立體結構不久,科學家們便開始了
蛋白質結構預測研究工作。這件事本身就可在一定程度上說明蛋白質結構預測工作的重要性。當時大多數的預測方法是依靠比對方法進行預測,而且直到目前為止對此問題還沒有完全解決,往往是某種方法對一類或是幾類蛋白質的二級結構的預測特別準確,而對其他幾類蛋白質的預測卻很不盡如人意。當然這其中有人們對整體蛋白質結構知識匱乏的因素,也有受限於當時的科學技術手段的因素。最早的比較成功的方法是Chou和Fastman在1974年提出了他們基於統計學的蛋白質二級交換結構預測的方法,之後又有很多人不斷地在改進算法以期提高預測的準確度。這其中有1978年Gamier,1987年Deleage和Roux,1996年King和Sternberg的SOPMA算法,1999年Guermeur的DSC算法等等,近年來很多方法已經不僅僅基於統計學的方法,兩是在原來統計學思想的基礎上增加了
多重序列比對、
神經網路、
SVM或是
決策樹的方法,其中最著名的就是1993年Rost和Sander的基於多重序列比對和多層神經網路的PHI)方法,蛋白質二級結構預測的準確度提高到70%,同時該方法也倡導了目前蛋自質二級結構預測中各種算法聯用的趨勢。
基本方法
生物信息學的主要目的之一在於了解蛋白質中胺基酸序列和三維結構之間的關係。如果知道了這種關係,就可以從胺基酸序列可靠地預測蛋白質結構。然而,序列和結構間的關係並不簡單。
目前有超過160000條蛋白質的序列可用,但其中僅有35343條蛋白質的結構是已知的(PDB資料庫2006-02.28的統計數據)。基因組的加速預計能加快解決蛋白質結構問題。蛋白質結構比較表明,新發現的蛋白質結構常常與已知結構具有相似的結構上的摺疊或構造。因此,
蛋白質摺疊成三維結構的許多方法是一致的。結構比較亦顯示出蛋白質中許多不同的序列在不同的結構環境中可以發現相同的短
胺基酸模式。二級結構中的胺基酸序列也被收錄在對結構預測有用的資料庫中。序列資料庫中許多蛋白質具有保守序列模式,這種保守模式可以進一步分類。
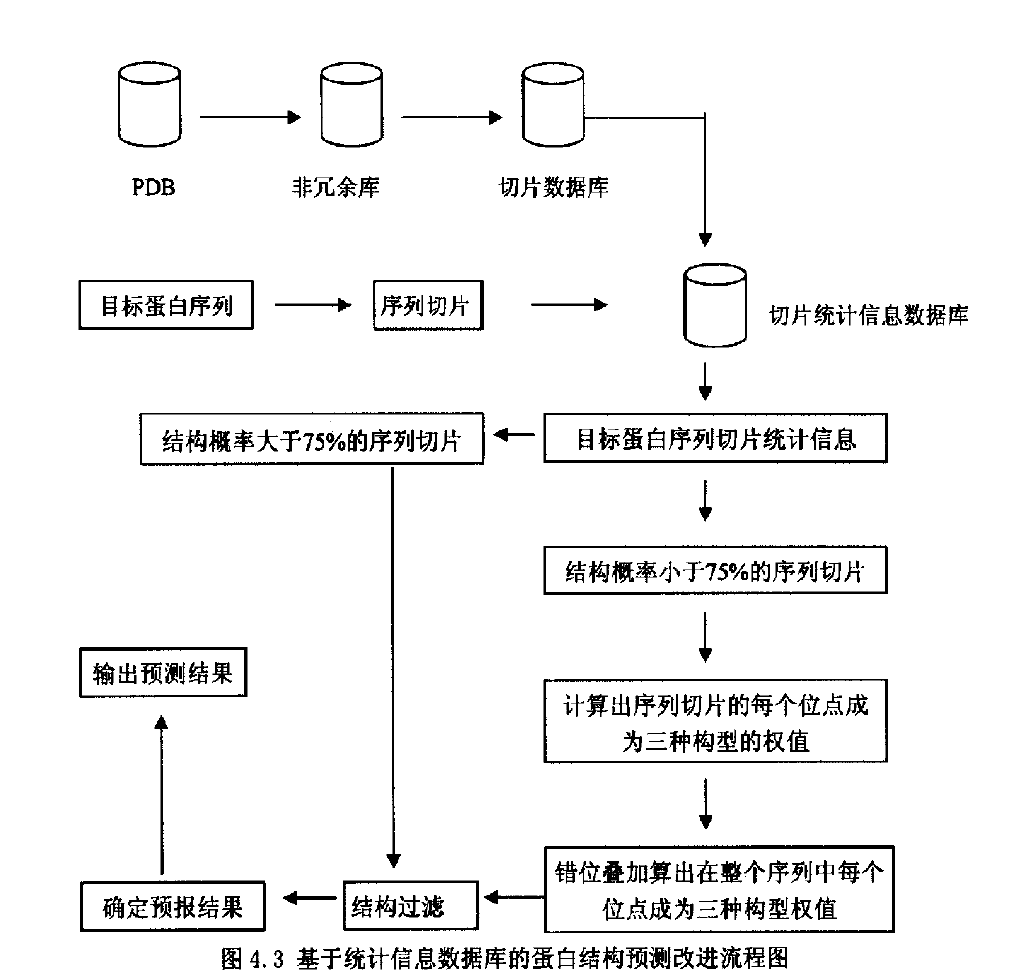
基於統計信息資料庫的蛋白結構預測改進流程圖
如果兩個蛋白質序列顯著相似,則它們也應有相似的三維結構。這種相似性可能體現在序列的長度上,或體現一個或多個已定位的區域中是否有相對短的模式。進行全局序列比對後,如果多於45%的胺基酸位置是相同的,則蛋白質三維結構中胺基酸的重疊性就高。因此,如果其中一個已比對過的蛋白質結構已知,則另一個蛋白質結構以及相同的胺基酸位置就可以可靠的預測。如果少於45%而多於25%的胺基酸相同,結構也可能相似,但在相應的三維位置上變異較多。為此,人們發展了大量的
蛋白質結構預測方法。
預測系統
目前基於蛋白質統計信息數據的預測系統的Web Server正在籌建中,由於整個系統構建的工作量是相當大的,故而暫時沒有發布。計畫中的Web Server將提供以下服務:
1.跟蹤PDB數據的數據更新,隨時更新統計信息庫中的數據;
2.提供蛋白質不同切片長度的結構機率及相關統計信息檢索服務;
隨著生物信息的發展,人們必將獲得越來越豐富的蛋白質結構數據,我們應該堅持利用數據挖掘這個有力的工具,對數據進行有效的處理,以期獲得更多有益的知識。
蛋白質綜合信息資料庫構建
蛋白質信息資料庫的建立是蛋白質結構研究的基礎,圍繞蛋白質的粗粒化結構及可能對蛋白質結構產生影響的因素,建立實用的非冗餘的資料庫是必要的。在非冗餘結構分類資料庫ASTRAL 的基礎上,將蛋白質的序列信息、二級結構信息、蛋白質亞細胞定位信息、功能信息、物種信息、PDB ID信息、SWISS.PROT ID信息集成,得到蛋白質綜合信息資料庫。其中PDB ID、SWISS.PROT ID信息來自Swiss.Prot資料庫,蛋白質序列信息、二級結構信息來PDB資料庫,
亞細胞定位信息來自DBSubLoc資料庫,結構類信息來自SCOP資料庫,蛋白質的物種信息信息取自PDBSOURCE,蛋白質的功能信息信息取自PDB RECORDS。
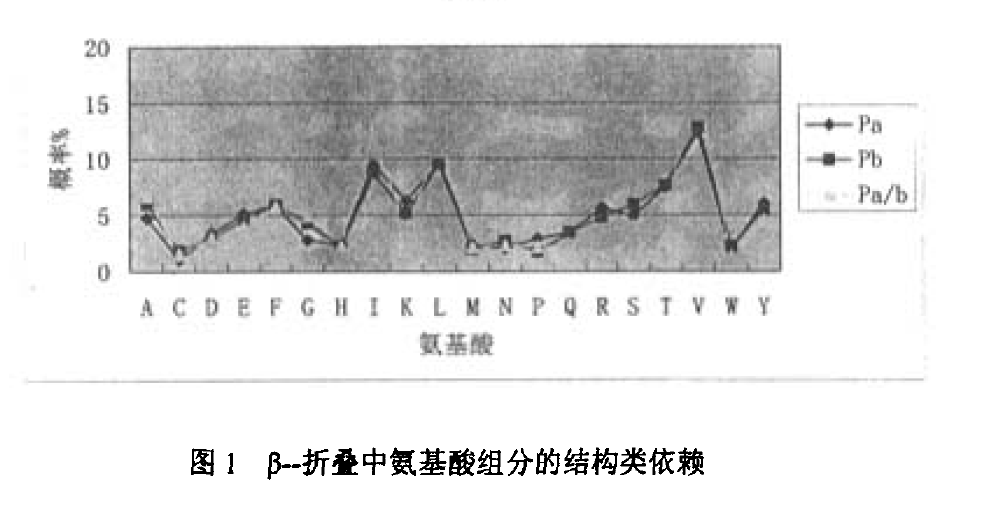
β一摺疊中胺基酸組分的結構類依賴