2010年, 美國科學家J. Craig Venter團隊報導了一種新興的含有一個人工合成具有1.08 M 鹼基基因組的支原體細菌。這個成就是化學家和生物學家協作努力通過五年研究出來的結果。初始細胞的生成需要很大的努力,一旦製得人工細胞,他們就會像自然細胞一樣增殖。因此,人工設計的細胞可以合理的成本再生,用作合成有用蛋白質和其他材料的生物工廠。現有的生物系統的重複設計是一個合成生物學途徑的例子。
另一合成生物學方法的類型是構建一個新的生物系統,用最新的生物組分來建造。在這個方法中,新的人造組分用於開發服務於某個目標,在生物系統中他們與側邊的自然組分一起運作。新的組分由重複的“proof of concept”試驗創建。原型組分基於某個概念或者主意設計,並根據物理和生物實驗結果進行改進。在這裡,我們通過對DNA遺傳密碼符號擴展的非自然鹼基對的創建,描述這種類型的生物合成方法。
基本介紹
- 中文名:非自然DNA鹼基對的分子設計
- 鹼基對:四個
- 包括:腺嘌呤(A),鳥嘌呤(G)等
- 例子:重複設計
自然鹼基對的發展,自然鹼基對: A-T和G-C,Benner團隊的親水性非自然鹼基對,結論,
自然鹼基對的發展
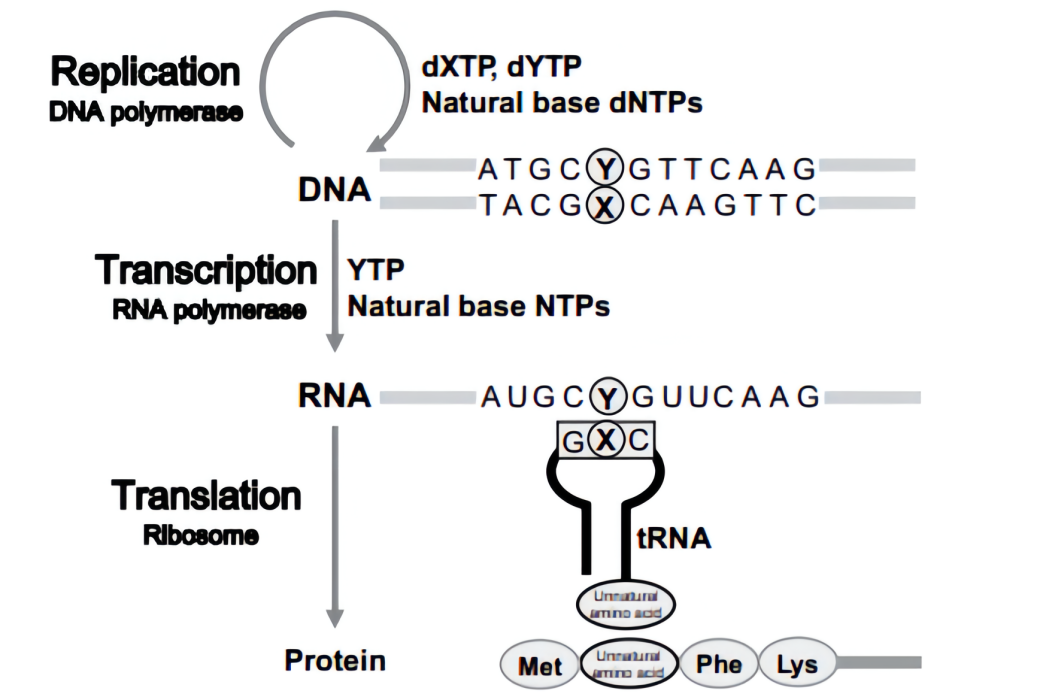
地球生命遺傳信息在DNA序列中有四個不同鹼基對編碼,腺嘌呤(A),鳥嘌呤(G), 胞嘧啶(C)和胸腺嘧啶(T)。DNA雙鏈分子中,A選擇性的與T配對,G與C配對。鹼基配對原則是基本的通過複製、轉錄和翻譯的遺傳信息流。因此,DNA中引入一個人為創建的鹼基對可以增加遺傳字母,擴大遺傳信息,提供一個新的生物技術能夠與特定的新組分結合為核酸和蛋白質(Figure 1)。9此外,最近的利用人工鹼基在複製和轉錄方面的研究已經顯示出新的分子相互作用和生物反應機理,僅僅通過自然組分利用生物分子標準進行饞鬼分子觀測不出來。而且,人為的提高生物系統遺傳密碼符號對於化學家來說是一個巨大的挑戰。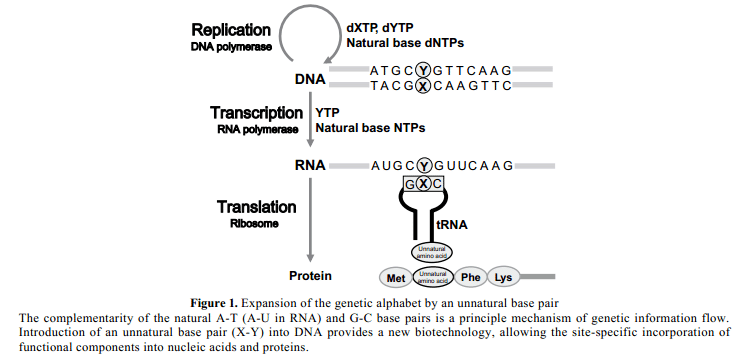

Figure 1
最重要的問題是,費自然鹼基對(X-Y) 作為三分之一的鹼基對具有很高的特有的選擇性,也就是說,X選擇性與Y配對,與此同時, A-T和G-C在生物系統中配對(Figure 2)。在複製中,DNA聚合酶結合底物與模板鏈之間部分雙鏈DNA片段。隨後,5'-三磷酸何乾(dNTP, substrate)進入DNA聚合酶絡合物。當基質在模板中與正確的夥伴配對,然後引物3'-羥基基團的氧原子進攻基板三磷酸α 位置。這個結果導致在引物和導入物之間形成磷酸雙酯鍵,並且釋放出焦磷酸做為一個離去集團。引物結合正確的基質後,DNA聚合酶滑向模板DNA 鏈,下一個基質結合發生。複製中自然鹼基配對的選擇性非常的高。例如,大腸桿菌DNA聚合酶I的克列諾片段,10,000 次結合會出現一次不正確的結合,10也就是說克列諾片段每一次複製的自然鹼基對選擇性為99.99%。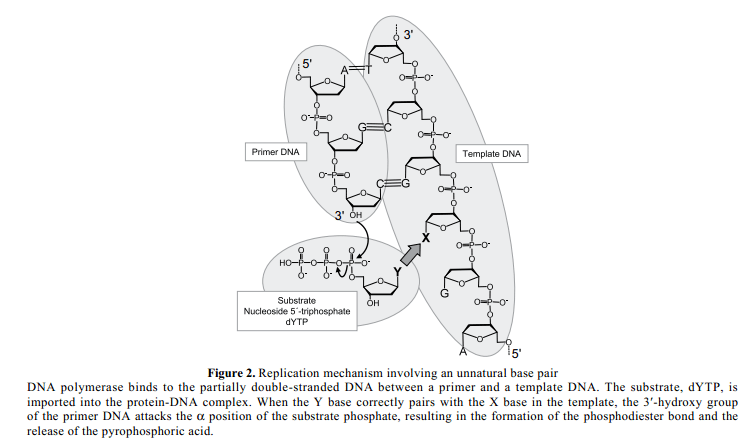

Figure 2
自然鹼基對: A-T和G-C
創建一個非自然鹼基對,我們需要了解為什麼A-T 和G-C 鹼基對按照他們的化學、物理、生物狀態在複製中有如此高的選擇性。在A-T和G-C 配對中,嘌呤鹼基(A or G)與嘧啶鹼基(T or C)配對, 兩個氫鍵用於A-T,三個用於G-C (Figure 3)。因此,每一個鹼基對糖苷鍵的距離約為10.7-11.0 Å,並且雙螺旋DNA 分子具有規則的人字齒輪結構。氫鍵在質子予體殘基和質子受體原子或殘基中間形成。A-T和G-C對之間,氫鍵模式各不相同,因此,A與T, G和C配對總是一種常規的螺旋結構。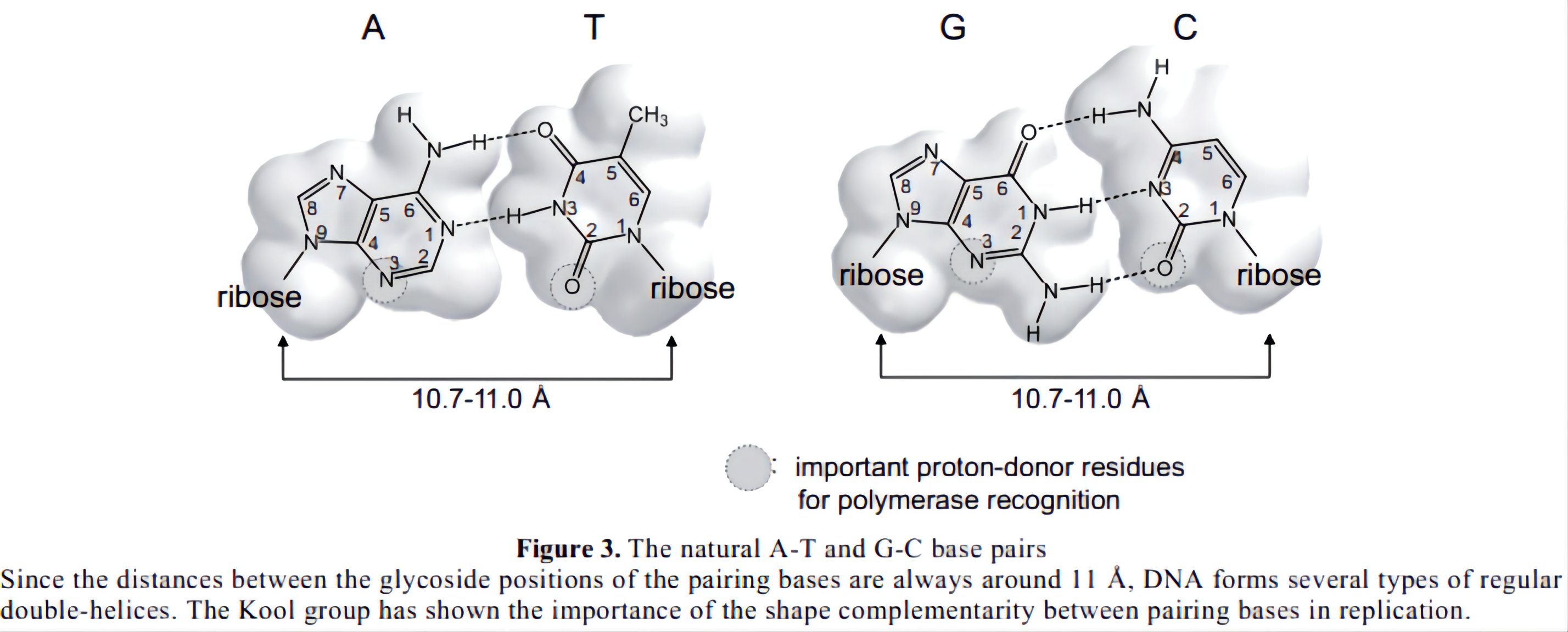

Figure 3
然而,最近的研究已經表明, 在複製中氫鍵的形成沒有必要搭配正確的鹼基對。
1995年,Eric Kool 和他的同事們設計合成了一個疏水性非自然Z–F對,其中Z和F 的形狀分別模仿A 和T(Figure 4)。11-13對於Z,A 中1- 和3-氮被碳替代,6-氨基被甲基替代。對於F,T中3-氨基被C-H替代,2-和4-酮基由氟原子替代。通常,一個氟原子質子受體的能力低於酮十倍。Kool團隊化學方法合成特定位置中含有Z 或F的 DNA 模板 ,以及他們的核苷三磷酸鹽(dZTP和dFTP),作為底物在體外進行複製實驗。他們表明,大腸桿菌DNA聚合酶I的克列諾片段在模板中有效的結合了dFTP 和dTTP 成為DNA ,對立與Z 或A,也與dZTP 、dATP 對立與F或T合併。相比之下,dCTP 相對Z 和 dGTP相對F的合併效率較低。因此,疏水性非自然Z-F對在複製中作用,與A-T對兼容,這表明,複製中配對鹼基之間形狀互補性重要於氫鍵互補性。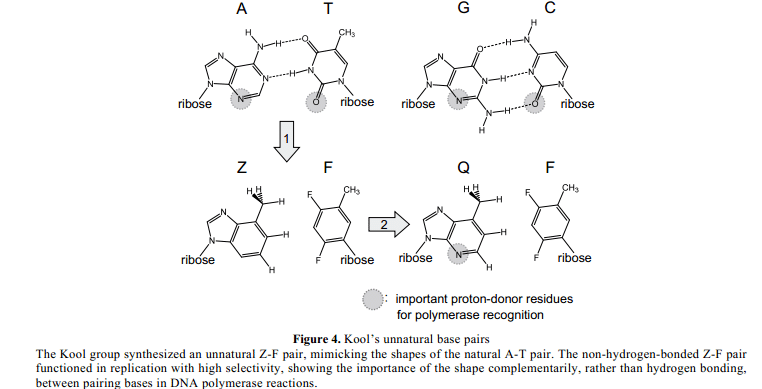

Figure 4
通過聚合酶來識別,A 和G中的3-氮, C和T中的2-酮作為氫鍵受體對於小溝是很必要的,在聚合酶中與具體的胺基酸殘基相互作用(Figure 3)。15例如,缺失2-酮基的嘧啶類似物基質在PCR中不能夠併入DNA ( P olymerase C hain R eaction).16Kool的Z鹼基相對於嘌呤3-氮缺少氫鍵受體原子,因此,他們新近研發Q 鹼基,其位置上有一個氮(Figure 4)。17克列諾片段被複製的Q-F鹼基對比Z- F對更高效。其他的因素,比如基質疏水性,基質偶極距,相鄰級之間堆積相互作用,以及CH- π相互作用18,在複製中對於鹼基對選擇性也很重要。
考慮到這些,目前為止非自然鹼基對的各種類型已經發展至此。下面的季節中么,我們將介紹具有代表性的非自然鹼基對以及他們的發展進程。
Benner團隊的親水性非自然鹼基對
在1980年代後期,Steven Benner和他的同事們報導了四種類型的具有不同氫鍵幾何體的 非自然鹼基對,這不同於A-T和G-C對。19, 20其中一個典型代表就是6-氨基-2-ketopurine (isoG)和2-氨基-4-ketopyrimidine (isoC)間的鹼基對,他們分別是G和C的結構異構體(Figure 5)。19Benner團隊證實了isoG-isoC對通過克列諾片段在複製中互補作用。此外,isoG基質(isoGTP)併入RNA 相對於isoC 併入DNA 模板,通過T7 RNA聚合酶。21此外,1992年,isoG-isoC對用於體外翻譯系統,通過一個短的化學合成mRNA(含isoC)和一個3-iodotyrosyl tRNA (含isoG) ,使得不標準胺基酸特定合併成為多肽。22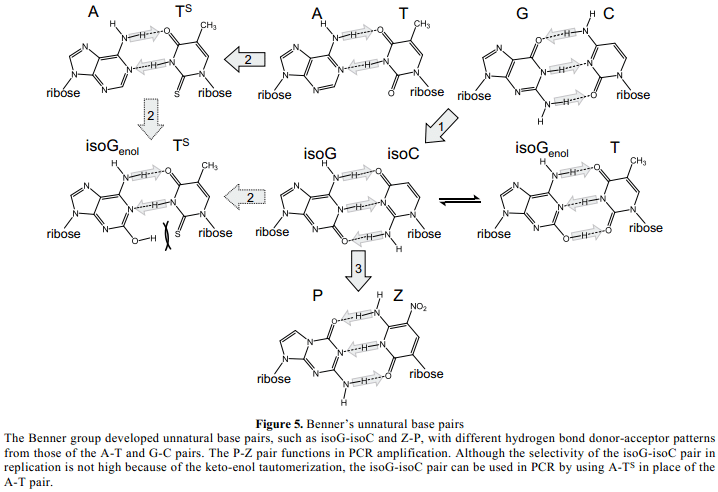

Figure 5
Benner團隊這些開創性的研究對利用非自然鹼基對的遺傳膨脹係數受到了很大的關注。然而,這些非自然鹼基對仍有很多的缺點。比如,isoG鹼基溶液中經受keto-enol互變異構化,isoG鹼基與T烯醇化(Figure 5)。21除了isoG的問題,isoC中的2-氨基,相當於自然鹼基的2-keto位置,降低與聚合酶的相互作用。21此外,isoC的核苷在水溶液中不穩定,室溫下,四天以後水中中性環境下會有50% 的isoC核苷三磷酸分解掉。23儘管在isoC鹼基位置3處(相當於自然嘧啶鹼基位置5處)引入甲基可以改良其穩定性,然而 甲基-isoC 核糖核苷仍然易受其β-糖苷鍵差向異構化作用,從β-轉換為α-形式。Benner團隊通過在位置3處引入一個硝基解決了這個問題,24如下所示。
2005年, Benner團隊通過利用一個改性的T 鹼基,2-thiothymine (TS),替代T (Figure 5), 解決了isoG keto-烯醇互變異構化的難題,用於isoG-isoC鹼基對系統。25TS中大尺寸的硫原子阻止了isoG形式中與烯醇的配對, 但仍是沒有空間斥力的配對。這個策略類似於Kool的配對鹼基鍵形狀適合概念,以及Harry 1988年報導的的非自然鹼基對的概念,26他利用一個改良的鳥嘌呤,6-thioguanine作為一個新的鹼基。非自然isoG-isoC對與A-TS對在酶聚合鏈反應中共同作用:isoG-isoC對的每一次複製選擇性達到98%,而isoG-isoC選擇性在沒有TS的幫助下每次複製達到93%。25然而,98%非自然鹼基對選擇性對於複製來說是不夠的,並且非自然鹼基對的保留率在PCR反應擴大DNA 片段的20周期中變為67% (0.9820 =0.67)。因此,對於複製中非自然鹼基對的實際使用中,每一次複製超過99% 的選擇性是必要的。
2007年,Benner團隊改進了非自然鹼基對系統,並研發了P-Z 對,其在PCR擴增中不需使用A-TS對(Figure 5)。24不同於isoG,P鹼基在溶液中不經歷不良的互變異構化。Z鹼基中的硝基基團由於其亞氨基的去質子化,阻止了它的核苷衍生物的差向異構化。雖然P-Z對選擇性當時在PCR中為97.5% ,但是最近的報導表明最佳化PCR 條件下改良的選擇性在99.8%。
Romesberg團隊的疏水性非自然鹼基對
Kool的關於非氫鍵鹼基對的開創性實驗引發疏水性鹼基對作為非自然鹼基對的候選物。Floyd Romesberg和他的同事開發了大量的疏水性非自然鹼基對,在複製系統中檢測他們的能力。1999年,他們報導了一個疏水性異喹啉衍生物PICS(Figure 6),其自我互補鹼基對在雙螺旋DNA 片段中具有很高的熱穩定性。28此外,PICS酶作用物與DNA酶結合通過克列諾片段與PICS在模板中相對應。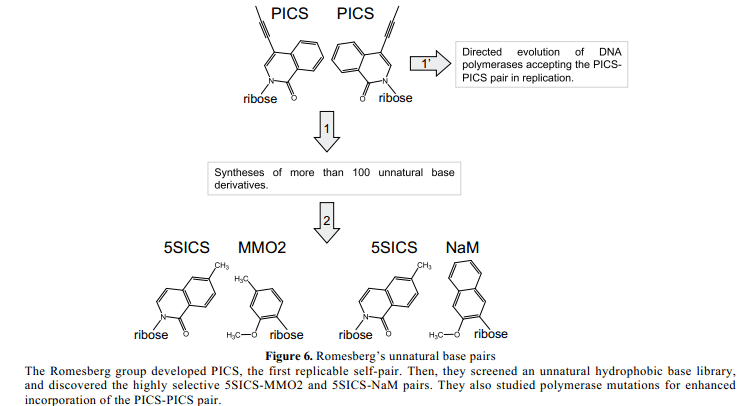

Figure 6
不幸的是,在PICS 基板中參入PICS後,引物暫停延伸。這是因為PICS-PICS對的形狀對於常規DNA雙螺旋的空間來說太大了,其PICS 鹼基相互堆疊,不允許聚合酶識別。因此,他們詳盡的設計和合成了另外的疏水性非自然鹼基對,並在複製中檢驗了它們的能力。
2009年,他們成功的開發了疏水性非自然鹼基對5SCIS-MMO2和5SCIS-NaM (Figure 6),他們的功能是作PCR擴增中三分之一的鹼基對。36, 37與之前的PICS–PICS對相比, 這些鹼基對的空間互補形狀有所改進。5SCIS–NaM 對在PCR中最佳的保真度達到99.8%,儘管它取決於周圍非自然鹼基對的排列順序。這些疏水性鹼基對通過T7 RNA 聚合酶在轉錄中也起作用。38該團隊還通過一個進化工程技術設計出DNA聚合酶,通過採用突變聚合酶了解決複製中使用PICS-PICS 鹼基對時聚合酶暫定問題。
Hirao團隊的一系列非自然鹼基對
我們團隊在1997年之後也研究了非自然鹼基對。大量的“proof of concept”實驗後,我們於201年開發出第一個非自然鹼基,在2-氨基-6-dimethylaminopurine (x)和2-oxopyridine (y)之間(Figure 7)。40這個x-y設計包含兩個概念:Benner氫鍵模式(它不同於自然鹼基對’s)和一個位阻效應。這個x-y對具有兩個氫鍵,x 的2-aminopurine 部分通過一個類似的氫鍵作用與T 配對。因此,我們在x的6位置處添加一個巨大的二甲胺基團, 通過T中6-dimethylamino 基團和4-keto基團之間的空間位阻來排除x-T配對。與 T相反, y 具有一個氫鍵代替了keto 基。x-y 對功能在轉錄中具有很高的選擇性,T7RNA聚合酶混合y三磷酸基質(yTP) 成為RNA,相對於x 在DNA模板中,具有高於95%的選擇性。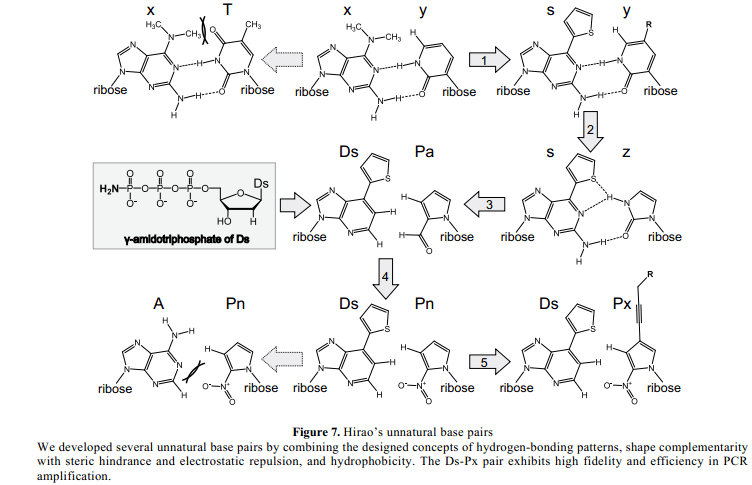

Figure 7
此外,我們通過用一個噻吩基替代二甲胺基改進了非自然鹼基對的形狀互補性。因此,我們開發了2-amino-6-thienylpurine (s)作為y的配對搭檔 (Figure 7)。41噻吩基的平面化比X二甲胺基更加有效的排除了與T 的錯誤配對。此外,平面性增加了s 鹼基與相鄰鹼基在DNA 鏈中疊加的能力。相比x-y 對,s-y對在轉錄中的選擇性和效率都有了很大的改進 不僅僅yTP 也包括yTPs的一些部分,通過T7 RNA聚合酶,比如biotin-和fluorophore-linked y 鹼基,與RNA結合,相對應於DNA模板中。42-442002年,通過結合特定的含有s–y對和一個E. coli體外翻譯系統的轉錄,我們成功的特定結合一個非標準胺基酸,3-chlorotyrosine變為一個蛋白質。41因此,s–y對可疑作為體外翻譯和轉錄系統中第三個鹼基 。
我們試圖進一步的改進我們這些非自然鹼基對。一個主要的障礙是s–y對複製中不足的選擇性。PCR的一個DNA含有s–y鹼基對片段10個周期過後,大約有40%的非自然鹼基對被自然鹼基對所替代。每一次複製中s–y對選擇性是~95%(40%的替代,10周期PCR過後: 1−0.9510=~0.4),因此,對於非自然鹼基對複製中超出99% 的選擇性時必須的,一個非自然鹼基對約~90% (0.9910=~0.90)可保留。因此,我們嚴格的精煉鹼基對之間的形狀互補,並設計了一個五元環核苷模型,imidazoline-2-one (z),45代替六元環y,作為s的配對搭檔 (Figure 7)。此外,我們祛除了來自s-z對之間的含有氫鍵相互作用的原子和殘基,46在7-(2-thienyl)imidazo[4,5- b]-pyridine (Ds)和pyrrole-2-carbaldehyde (Pa)之間開發了一個疏水性非自然鹼基對 (Figure 7, 參閱化學合成附錄)。47我們在Pa 鹼基上添加一個醛基,作為一個質子受體用於聚合酶之間的相互作用。
Ds–Pa對的問題是self-complementary Ds-Ds配對在複製中也有競爭力的發生。也發現了Romesberg’s PICS–PICS對,在Ds合併含有 對立面的Ds時的擴展停頓。幸運的是,我們發現了一個改良過後的Ds三磷酸基質,γ-amidotriphosphate, 選擇性的結合opposite Pa,但是結合opposite Ds之後顯示出大幅度的減少。47改進過後的基質合併,因為基質的amidopyrophosphate部分被祛除作為一個聚合作用中的離去集團,DNA具有母磷酸二酯鍵合。為了PCR擴增,我們除了Ds也使用A中的γ –amidotriphosphate,來降低dATP 相對Pa.的插入性錯誤。2006年,我們使用A中的γ –amidotriphosphate和Ds,成功的完成了含有Ds–Pa對的高選擇性PCR擴增,其中每一次複製Ds–Pa對的選擇性達到99%以上。一個偶然的失誤,我們通過處理三磷酸合成中間體意外的合成了γ -amidotriphosphates,5'-三磷酸酯環狀物,具有濃縮氨。
我們進一步的改進了Ds-Pa對,用硝基替代Pa中的醛基,因此設計了2-nitropyrrole (Pn)(Figure 7)。48Pn的硝基通過硝基中的氧和A中的1-氮之間的靜電斥力可以減少A opposite Pa的插入性錯誤。另外,我們在Pn鹼基4-位置處添加丙炔基,開發出 2-硝基-4-propynylpyrrole (Px) 作為Ds的配對搭檔(Figure 7)。49丙炔基增加了疏水性,增強了聚合酶之間的相互作用。因此,Ds-Px對在PCR中的功能不需要γ –amidotriphosphates的援助。含有Ds-Px對的DNA 片段通過PCR 40周期後被放大108-fold,超過97%的Ds–Px對仍被保留在擴大的DNA中。The selectivity of the Ds–Px對的選擇性在每次複製中達到99.9%以上,這是迄今為止非自然鹼基對中最高的選擇性。50