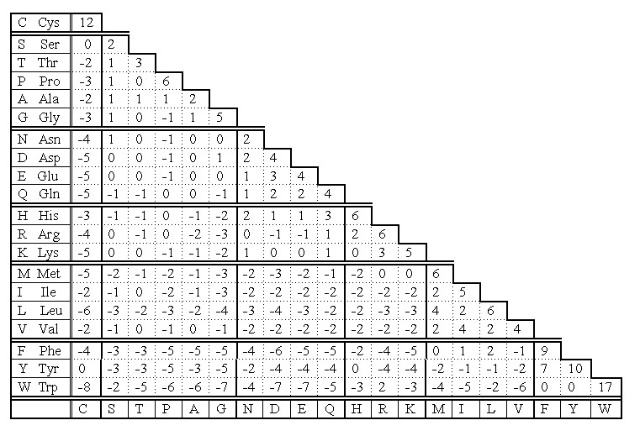
PAM打分矩陣是生物信息學中用來判斷兩個蛋白質序列相似性的矩陣,這個矩陣的各個格點上的數值為這一胺基酸被其他胺基酸替換的機率。
基本介紹
- 中文名:PAM打分矩陣(Dayhoff突變數據矩陣)
- 外文名:Point Accepted Mutation Matrix
- 原理:基於進化的點突變模型
PAM矩陣(和BLOSUM矩陣)都是用於蛋白質序列相似性的記分矩陣(scoring matrix)。記分矩陣中含有對齊時具體使用的數值。一般FASTA和BLAST都提供BLOSUM或PAM系列矩陣供選擇,若要進行突變性質的進化分析時可以使用PAM,FASTA預設推薦BLOSUM矩陣。
PAM (Point Accepted Mutation)矩陣基於進化原理,建立在進化單點可接受突變(PAM)模型基礎上。如果兩種胺基酸替換頻繁,說明自然界接受這種替換,那么這對胺基酸替換得分就高。一個PAM就是一個進化的變異單位,即1%的胺基酸改變,但這並不意味100次PAM後,每個胺基酸都發生變化,因為其中一些位置可能會經過多次突變,甚至可能會變回到原來的胺基酸。PAMn矩陣是由PAM1自乘n次得到的。
PAM (Point Accepted Mutation)矩陣基於進化原理,建立在進化單點可接受突變(PAM)模型基礎上。如果兩種胺基酸替換頻繁,說明自然界接受這種替換,那么這對胺基酸替換得分就高。一個PAM就是一個進化的變異單位,即1%的胺基酸改變,但這並不意味100次PAM後,每個胺基酸都發生變化,因為其中一些位置可能會經過多次突變,甚至可能會變回到原來的胺基酸。PAMn矩陣是由PAM1自乘n次得到的。
這種矩陣的缺點是一旦PAM1的矩陣有誤差,那么自乘250後得到的PAM250矩陣的誤差就會變得很大。這個矩陣現在已經很少使用,而被BLOSUM代替。